理化学研究所 生命機能科学研究センター 先端バイオイメージング研究チーム チームリーダー
細胞に光を当てると、光は細胞の内部で屈折や反射を繰り返しながら出てきます。そのため、細胞を通った光には、細胞内の情報が含まれています。
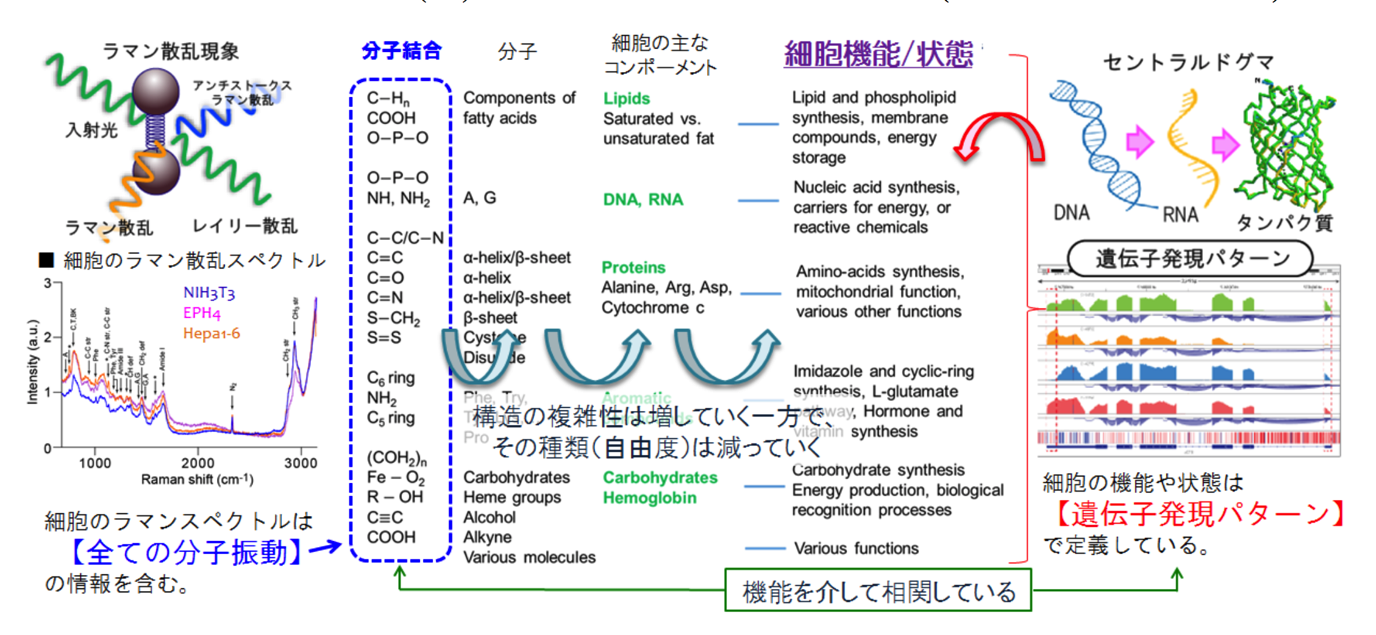
散乱光にはいくつもの種類がありますが、ラマン散乱は、分子構成がもつ固有のエネルギー(分子振動)によって色が変わります。たとえばC=CとR−OHはそれぞれ異なるエネルギーを持っているため、C=Cに応じた色、R−OHに応じた色が生じるのです。細胞の分子構成が変化すれば、ラマン散乱の色も変化します。
このようにラマン散乱スペクトルは、細胞を構成するすべての分子の情報を網羅しています。ある特定の分子は、特定のラマンスペクトルを示すので、スペクトルのピークを解析することで、細胞の分子構成を解析できるのです。しかし実際は、これらの情報が非常に複雑であるため、すべてを解析することは困難です。
私はこの複雑なラマン散乱スペクトルから、細胞の分子構成ではなく、遺伝子発現を予測できるのではないか、と考えました。
細胞のラマン散乱スペクトルデータとRNAシーケンスデータを収集し、対データの相関関係を明らかにすることで、ラマン散乱スペクトルデータからRNAシーケンスを推定する機械学習/深層学習モデルを構築します。
そのために、まず100種類のiPS細胞を用意し、明視野画像とラマン散乱スペクトルデータを収集します。1種類のiPS細胞に対して数千個の細胞を計測するため、顕微鏡の自動制御システムを開発してデータ収集にあたります。
次に、ラマン散乱スペクトルデータを取った直後のiPS細胞を溶解させ、RNAシーケンス解析を行ってデータを取得。部分最小二乗法(PLS)を基本とした機械学習モデルでRNA発現パターンを予測し、直接的に寄与するラマン散乱スペクトルピークを明らかにして「ラマン散乱スペクトルから遺伝子発現を予測する機械学習モデルの開発」に繋げます。
まずは実験系を確立し、3カ月かけてフィージビリティスタディ(実現可能性の検証)を行いました。
 ラマン散乱光により遺伝子発現を予測/推定できる技術を開発する
ラマン散乱光により遺伝子発現を予測/推定できる技術を開発するラマン散乱スペクトルが表す分子振動の比率は、細胞の分子種の比率によって決まります。分子種の比率を決めるのは、細胞の種類や機能です。
細胞の種類や機能を決定するのは、遺伝子発現パターンです。このパターンが変化すれば、細胞の分子種の比率が変化し、ラマン散乱スペクトルも変化します。
このように、遺伝子発現とラマン散乱スペクトルは、超複雑系である細胞の表現系を介して、間接的に相関関係があると言えます。
遺伝子の種類と細胞の分子種には大きな差があり、数学的には相関を表すことは不可能ですが、先ほど述べたように細胞の遺伝子発現パターンは270種類まで減少するため、相関を見いだすことは十分可能です。
実際、1年目の研究では11種類の薬剤耐性大腸菌株を対象として、それぞれのラマン散乱スペクトルとRNAシーケンスの対データを収集し、相関をクラスタライズすることによって、ラマン散乱スペクトルと遺伝子発現に相関があることを実証しました。それ以前の研究でも、ラマン散乱光による「細胞の種類を区別する」「細胞の成熟度を評価する」計測技術を開発しており、コンセプトは証明できたと考えています。
 iPS細胞とRNAシーケンス解析装置は筑波の研究所にあり、2施設の協働で研究は進められた
iPS細胞とRNAシーケンス解析装置は筑波の研究所にあり、2施設の協働で研究は進められた