

2007年東京大学工学部卒業。東京大学大学院総合文化研究科広域科学専攻修士課程と生産科学技術研究所に所属、2013年にカリフォルニア大学バークレー校機械工学科博士課程卒業。2014年より東京大学大学院理学系研究科助教。同年に科学技術振興機構さきがけ兼任研究者、2015年には科学技術振興機構さきがけ専任研究者となる。2016年、シンクサイト株式会社設立。2018年より、東京大学先端科学技術研究センター准教授。
様々な分野の技術を融合して、新しいバイオテクノロジーの提案と開発を行っています。
具体的には、光学やマイクロ流体などの材料技術、生物、化学分野の技術を組み合わせ、情報技術も駆使します。これまでにない解析のプラットフォームを創出し、生命科学や医学の進歩に資することを目指しています。
細胞の解析には、様々な装置を使います。もし、異なる装置で読めるバーコードを細胞に付与できれば、異なる装置で得られた細胞データを紐付けできる。それによって各データの価値が高められると考えました。
「光核酸バーコード」は、その構想を具体化したものです。着色した微細なビーズを、細胞と一緒にゲルのカプセル中に閉じ込めます。ビーズは、色ごとに異なる核酸(DNA)が化学修飾されており、ビーズの色とDNAの塩基配列がバーコードとしての役割を果たしています。
顕微鏡では、ビーズの蛍光色が見えます。シーケンサーではビーズの色は観察できませんが、DNAの塩基配列を読み取ることができます。シークエンス解析の手前のアッセーで、各細胞の核酸とDNAバーコードを一緒に束ねる工夫を行うことで、シークエンスデータの中で紐付けが行えるようにしています。こうして、顕微鏡とシーケンサーで得られたデータが一対一で対応する、という仕組みです。
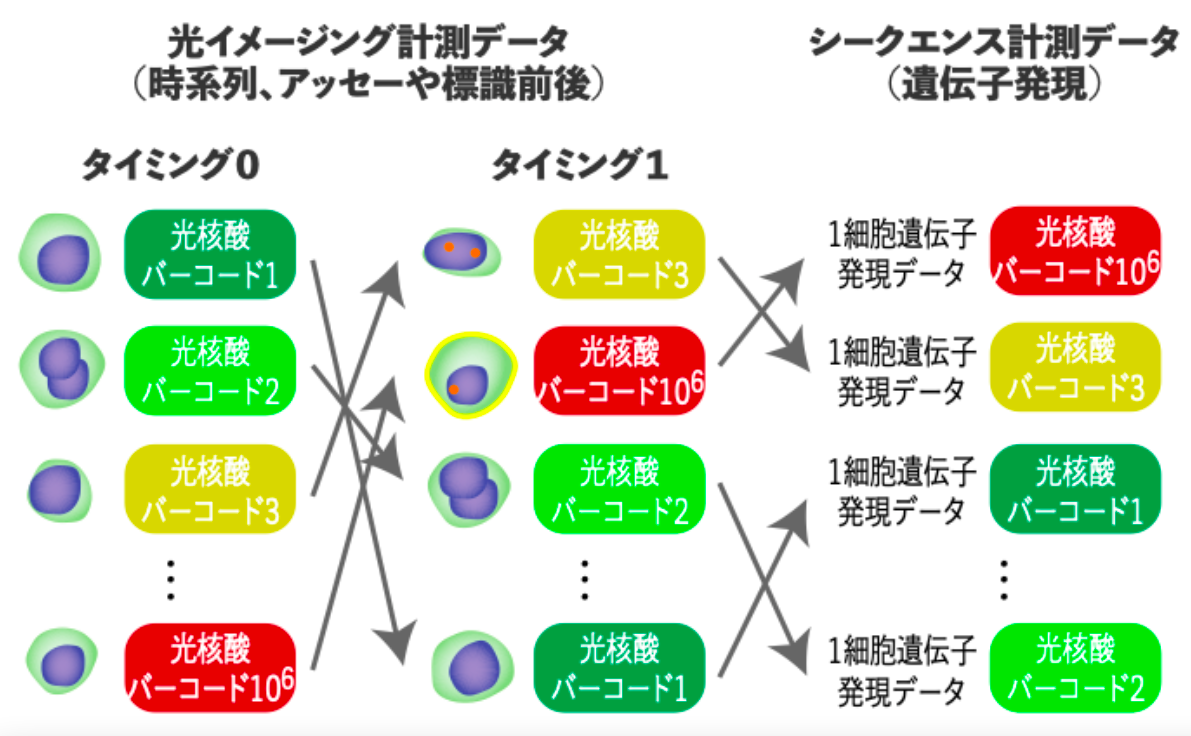 光核酸バーコードビーズにより、時間や装置間のギャップを越えて、細胞レベルでの計測データの紐付けを可能に
光核酸バーコードビーズにより、時間や装置間のギャップを越えて、細胞レベルでの計測データの紐付けを可能にその通りです。しかし、例えば10万個の細胞を観察したいからといって、10万種類のビーズを作るのは、現実的ではありません。そこで、ビーズの種類は数十種類とし、それらをランダムに組み合わせることにしました。このトリックを仕込むことで、解析スケールを100万細胞程度まで上げることを目指しています。
このバーコードは、細胞の時系列変化を追う際にも有効です。細胞の位置が変わっても個々の変化を正確に追うことができるためです。フローサイトメトリーのように、大量の細胞を溶液中に流しながら観察する場合にも対象をバーコードでトラックできるため、特に威力を発揮します。
 バーコーディング技術により様々な情報が紐付けられたデータベースをAIに学習させれば、まだ知られていない細胞の挙動や性質まで解き明かすモデルを作れると期待している
バーコーディング技術により様々な情報が紐付けられたデータベースをAIに学習させれば、まだ知られていない細胞の挙動や性質まで解き明かすモデルを作れると期待している