具体的には、どのような仕組みで染色体がたたまれているのですか。
高次構造の形成には、物理的な特性が大きく影響します。DNAはヒストンというタンパク質に巻きついているので、染色体そのものには硬さがあります。一方で、タンパク質どうしが親和性を持つ部分もある。一例としては、親和性の高い2箇所のタンパクがリング状の狭い空間を同時に通ることでくっついて離れなくなる、という折りたたみのメカニズムがあることがわかっています。
さらに、核の一部は粘稠性が高く、染色体が自由に動けるわけではありません。様々な制限の中で、動ける部分は動くし、動けないものは動かない。物理的な法則によって、染色体どうしがコンタクトできる場所とできない場所が、必然的に決まってくるのです。
物理的な法則が高次構造を作るのですね。では、そうやって折りたたまれた染色体は、どのように遺伝子の発現を制御しているのですか。
Hox遺伝子群という一連の遺伝子群の発現を観察したところ、遺伝子が、並んでいる順に発現していく様子が確認できました。その実験データと照らし合わせて、核の中には転写が抑制されている「ネガティブ」な領域があり、ネガティブな領域にまとめられている染色体が引きずり出されると、その領域から出た部分はアクティブになって遺伝子が順に発現してゆく、というメカニズムを考えました。
後に、他の研究者から出された結果や、古関明彦先生との共同研究で、核の中に「ポリコムボディ」と呼ばれるポリコムタンパク質群とDNAからなる巨大な複合体が形成され、DNAがそこから出ると発現され、入ると抑制される、というかたちで、遺伝子の発現のON・OFFが切り替わることが確かめられました。
染色体はダイナミックに動いていて、しかもそれが遺伝子の発現と抑制を反映していることがわかってきたのです。
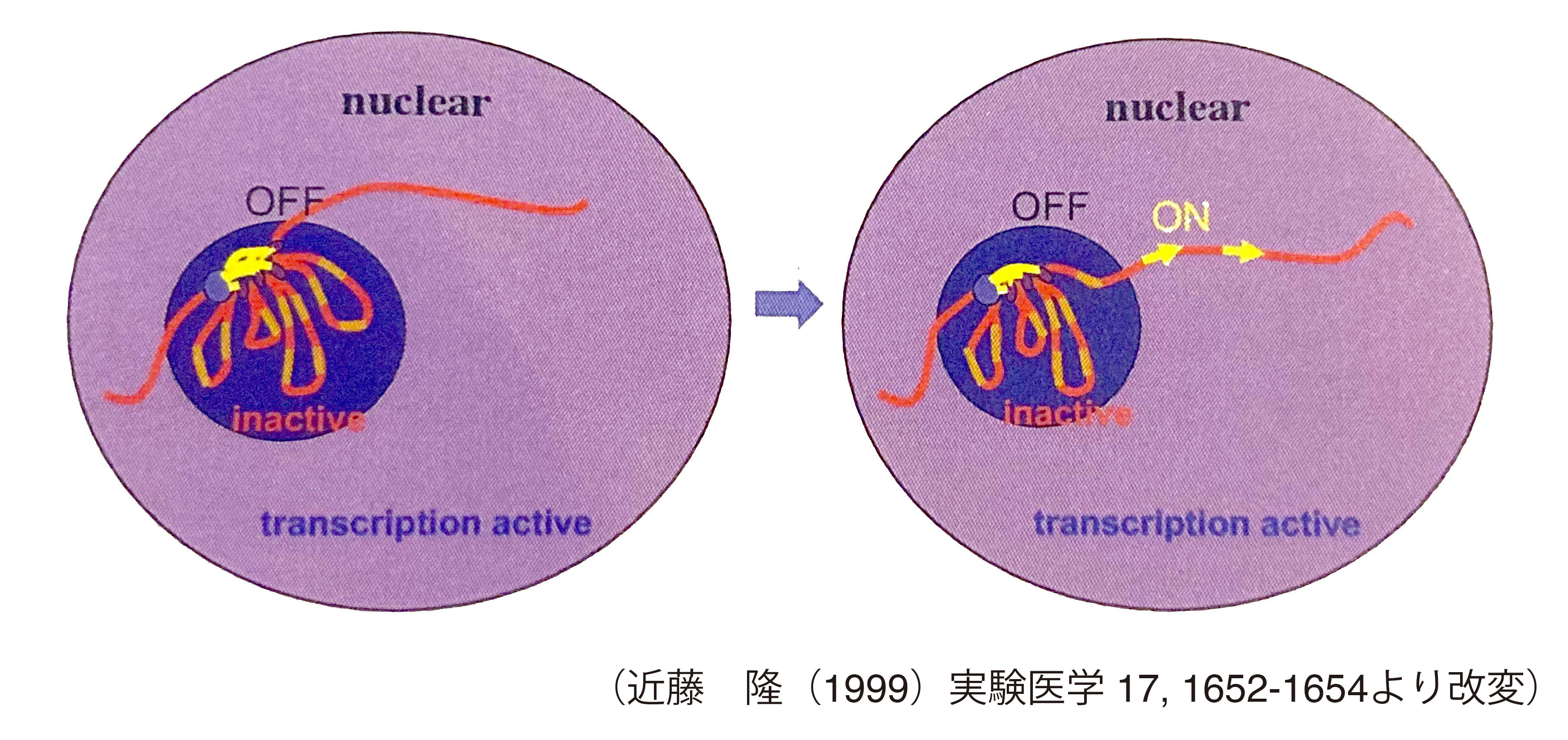
染色体が、核の中にあるネガティブな領域からポジティブな領域に引き出される様子。ネガティブな領域では、転写が抑制されている
それでは、エンハンサーとプロモーターが、ネガティブな領域から引き出されてアクティブになれば、遺伝子が発現するということでしょうか。
発現すべき遺伝子のエンハンサーがアクティブになることは必要ですが、プロモーターと離れてアクティブになっても意味がありません。エンハンサーがプロモーターを引き寄せたうえでアクティブになると、初めてプロモーターがアクティブになるのです。
さらに、発現を開始するプロセスだけでなく、止めるプロセスもあります。その役目を担うのが「サイレンサー」で、サイレンサーがつくとプロモーターの転写が止まります。
このように、複数のメカニズムが重なってようやく一つの遺伝子の発現と停止が適切に行われます。染色体の高次構造そのものが、発現を制御するセーフロックにもなっているともいえるでしょう。
染色体が動いている様子は、実際に観察できるのですか。
「FISH」という手法を用いると、染色体の挙動を時系列で捉えることができます。
遺伝子に結合するDNAフラグメントを作って、例えばプロモーターを青、エンハンサーを赤、サイレンサーを緑で標識します。すると、初めは緑と青が重なっていて、赤が離れています。プロモーターとサイレンサーがネガティブな領域で接触していて、エンハンサーが外に出ている状態です。それから一定の時間が経つと、3色が重なっていることが観察されました。この時期にはまだ遺伝子の発現はしていません。やがて発現が始まると、青と赤がくっついて、緑が離れている。すなわち、サイレンサーだけがネガティブな領域に残されていると考えられます。
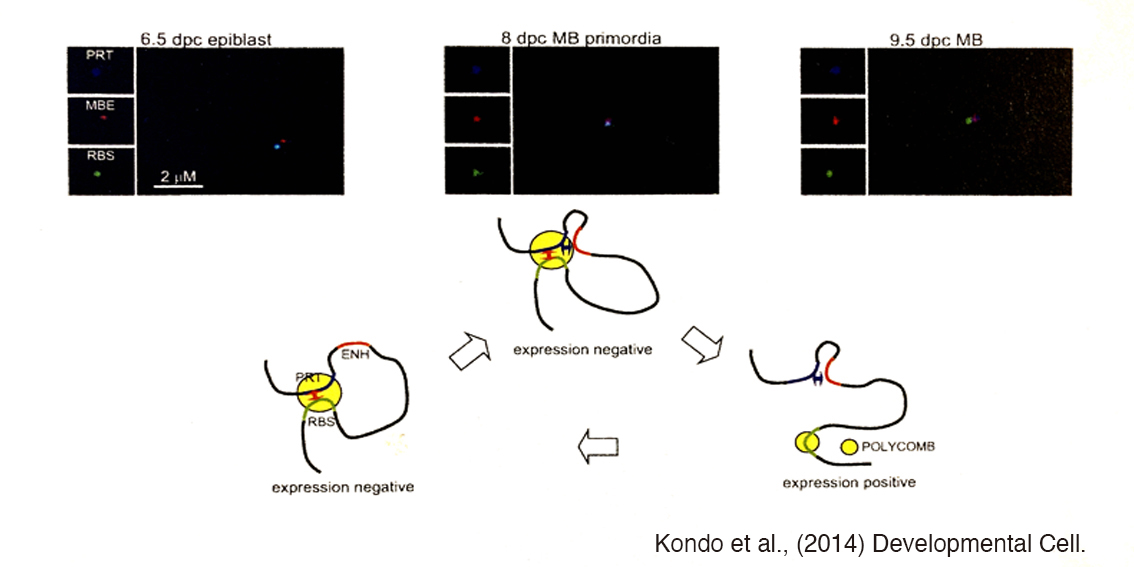
FISHで得られる映像では各色のプローブが光り、プロモーター、エンハンサー、サイレンサーの挙動を追うことができる


 染色体が、核の中にあるネガティブな領域からポジティブな領域に引き出される様子。ネガティブな領域では、転写が抑制されている
染色体が、核の中にあるネガティブな領域からポジティブな領域に引き出される様子。ネガティブな領域では、転写が抑制されている FISHで得られる映像では各色のプローブが光り、プロモーター、エンハンサー、サイレンサーの挙動を追うことができる
FISHで得られる映像では各色のプローブが光り、プロモーター、エンハンサー、サイレンサーの挙動を追うことができる